Chronic Illness
Diagnosis Helper: proposal of a tool to aid the diagnosis of
chronic diseases through the historical analysis of symptomatic reports
Michael Lopes Bastos 1 https://orcid.org/0000-0001-6581-2067
Anthony Lins 2 https://orcid.org/0000-0002-7153-841X
1 Escola Politécnica de Pernambuco, Universidade de
Pernambuco, Recife, Brasil,
2 Departamento de Jogos Digitais, Universidade Católica
de Pernambuco, Pernambuco, Brasil,
E-mail do autor
principal: Michael Lopes Bastos mlb@poli.br
Resumo
Segundo dados da
Organização Mundial da saúde (OMS), as doenças crônicas não transmissíveis
(DCNT) são responsáveis por cerca de 71% dos óbitos em todo o mundo. Desse
modo, ao longo dos anos algumas medidas vêm sendo tomadas para tentar reduzir
esse índice. No que diz respeito ao uso de tecnologias nesse processo, existem
algumas iniciativas no contexto do Aprendizado de Máquina (AM) que tentam
encontrar formas que vão desde o auxílio ao diagnóstico até o suporte em determinados
tipos de tratamentos. Visando isso, esse projeto tem como intuito apresentar
uma ferramenta, baseada em um modelo de aprendizado de máquina, para auxiliar
profissionais da saúde no diagnóstico das DCNT usando dados sintomáticos
derivados da base “Chronic illness” da plataforma Kaggle. Como
melhor resultado desse processo, foi escolhido um modelo de aprendizado baseado
em técnicas de ensemble, onde a melhor precisão obtida chegou a ≈ 71,63
% para um número de 20 patologias, sendo esse modelo usado como base para a
aplicação Chronic Illness Diagnosis Helper (CIDH), desenvolvida para uma
prova de conceito inicial.
Palavras-Chave: Doenças Crônicas; Inteligência
Artificial; Auxílio ao diagnóstico;
Abstract
According
to data from the World Health Organization (WHO), the noncommunicable chronic
diseases (NCD) are responsible for around 71% of deaths in all world. Thus,
over the years some methods have been taken to try to reduce this index.
Concerning to use of Technologies in this process, there are some initiatives
in the context of Machine Learning (ML) that trying to find ways from diagnosis
aid to support in certain types of treatments. Thus, this project has a goal to
show a tool based on a machine learning model to health professionals to
diagnosis NCD using symptomatic data derivates from base “Chronic illness” from
the Kaggle platform. As the best result from this process, was choose a
learning model based an ensemble technics, when the best accurate arrived at ≈
71,63 % for some 20 pathologies, being this model used as bases to the
application Chronic Illness Diagnosis Helper (CIDH), developed with an initial
Prove of Concept.
Key-words: Chronic diseases; Artificial Intelligence; Diagnostic aid.
1
Introdução
As
doenças crônicas ou doenças crônicas não transmissíveis (DCNT) segundo o estudo
“Saving lives, spending less: a strategic response to NCDs” feito em 2018
pela Organização Mundial da Saúde (OMS), são responsáveis pela morte de cerca
de 41 milhões de pessoas por ano, correspondendo a aproximadamente 71% dos
óbitos da população mundial. Dentre todas essas ocorrências, 15 milhões são de
pessoas com idade entre 30 e 69 anos e mais de 85% dos casos ocorrem em países
de média e baixa renda mundial [1].
No
Brasil, segundo o Ministério da Saúde, em 2015 essas doenças foram responsáveis
pela morte de 51,6% da população que possuía de 30 a 69 anos de idade. De uma
forma geral, as DCNT são chamadas de multifatoriais, ou seja, são acarretadas
por uma série de fatores, sejam eles sociais ou individuais. A grande maioria
dessas doenças possuem quatro fatores de risco em comum, são eles: o tabagismo,
atividade física insuficiente, alimentação não saudável e o uso nocivo do
álcool [2].
No que
diz respeito ao uso de tecnologias nesse processo, existem algumas iniciativas
no contexto do Aprendizado de Máquina (AM) que tentam encontrar formas que vão
desde o auxílio ao diagnóstico até o suporte em determinados tipos de
tratamentos [3]. Entretanto, a grande maioria dos estudos
são voltados a doenças crônicas específicas, como doenças cardiovasculares [4], doenças arteriais coronárias [5] e
doenças renais crônicas [6], por exemplo. Todos esses projetos aplicam
técnicas de Inteligência Artificial (IA) como método base para chegar as
soluções pretendidas.
Contudo,
ainda existem muitas lacunas que podem ser preenchidas no que diz respeito ao
uso das técnicas de IA no contexto das DCNT. O tratamento e diagnóstico médico
ainda enfrentam uma série de dificuldades em relação a análise do grande volume
de dados que existem atualmente. Os prejuízos e malefícios que as doenças
crônicas trazem à saúde das pessoas é algo preocupante e aumenta continuamente [3].
Dessa
forma, esse projeto tem como objetivo desenvolver uma ferramenta, baseada em um
modelo de aprendizado de máquina, para auxiliar profissionais da saúde no
diagnóstico das DCNT, tendo como base relatos sintomáticos de pacientes.
2
Métodos
Para o
desenvolvimento do projeto, a base escolhida foi a “Chronic illness:
symptoms, treatments and triggers” disponível no site Kaggle, uma
comunidade online de cientistas de dados gerenciada pela Google LLC. Esse
conjunto de dados é alimentado por informações cadastradas de pacientes com
diferentes tipos de doenças crônicas em diferentes países do mundo. A coleta é
feita a partir da plataforma Flaredown, um sistema voltado para obtenção
de informações de sintomas, alimentação, clima, medicamentos e tratamentos de
pessoas com esses tipos de patologias.
O
processo metodológico desse trabalho é baseado em conjunto de três módulos,
cada um possuindo uma série de passos sequenciais e iterativos, fazendo uso das
técnicas e métodos necessários para chegar até a versão final da ferramenta
proposta. A divisão dos módulos é feita de acordo com a natureza do processo,
onde a primeira etapa é denominada Manipulação de dados, por possuir as
atividades principais relacionadas a manipulação das informações da base de
dados escolhida. O segundo módulo é descrito como Definição da ferramenta,
pois trata de todo o processo de concepção e ideação para o desenvolvimento da
prova de conceito (do inglês Proof of Concept - PoC) sugerida para o
trabalho. A última etapa do processo é intitulada Desenvolvimento, pois
lida com a parte da implementação dos conceitos definidos nas etapas
anteriores. A Figura 1 demonstra um fluxograma geral desses módulos e
etapas do processo.
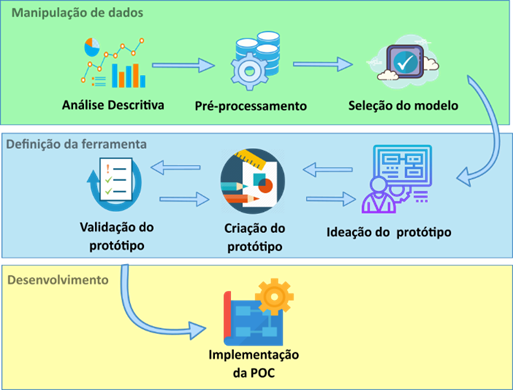
Figura 1: Processo metodológico para concepção final
da ferramenta. Fonte: Próprio autor.
O
módulo de Manipulação de dados é iniciado com a realização da Análise
descritiva das informações da base de dados. Essa etapa é realizada para
entender melhor como os dados estão dispostos e quais as abordagens devem ser
levadas em consideração na etapa seguinte, o pré-processamento das informações.
Esse ponto da fase de manipulação é um dos mais delicados e demorados do
módulo. Nele, é feita uma reestruturação de várias colunas da base, remoção de
valores inválidos e, seleção dos campos considerados mais importantes para o
processo de classificação.
Visando
a construção de um bom classificador, duas atividades complementares são desenvolvidas.
Primeiro são implementados modelos de aprendizado de máquina convencionais, com
o KNN e MLP. Depois é realizado um Ensemble com um conjunto heterogêneo de
classificadores (etapa de seleção de modelos). Ao fim, os resultados são comparados
e o melhor modelo é definido.
Após a
seleção do modelo, é iniciada a etapa de Definição da ferramenta. Nesse ponto é
realizada a ideação, criação e validação do protótipo do sistema.
Tendo
a base da aplicação criada e validade, é dado início ao processo de
implementação, que é a união do modelo de aprendizado de máquina criado com a
codificação do protótipo concebido no módulo de Definição da ferramenta.
3
Resultados
Para o
desenvolvimento desta seção, serão abordados os resultados obtidos em cada uma
das etapas do processo metodológico. Da subseção 3.1 até a 3.3 serão abordados
os aspectos referentes a etapa de manipulação de dados. A subseção 3.4 relata
as informações referentes ao processo de definição da ferramenta. Por fim, a
subseção 3.5 explica a etapa de criação da prova de conceito final
3.1
Análise Descritiva
Para
melhor entender as informações contidas da base “Chronic
illness”, foi necessário fazer uma exploração descritiva das
informações existentes. Inicialmente, a base continha um conjunto de 9 colunas e
3.487.633 registros armazenados. O Quadro 1
representa a descrição de cada uma das colunas contidas na base.
Quadro 1: Descrição das características principais da base
|
Coluna Descrição
|
|
user_id
|
Se refere ao identificador único dos
pacientes, armazenado em um formato de string.
|
|
age
|
Relata a idade de cada paciente.
|
|
sex
|
Retrata um conjunto de quatro
valores para sexo: “female”, “male”, “other” e “doesnt_say”.
|
|
country
|
Descreve a sigla do país ao qual o
paciente está imputando a informação. Ex.: ‘BR’
|
|
checkin_date
|
Registra a data no formato
AAAA-MM-DD em que o paciente registrou a informação.
|
|
trackable_type
|
Se refere a 7 tipos diferentes de
informações inseridas pelo usuário, são elas: Symptom (1.571.134
amostras), Weather (627.480 amostras), Condition (505.461
amostras), Treatment (383.201 amostras), Tag (207.902 amostras), Food
(192360 amostras) e HBI (95 amostras). O tipo Condition se refere a
patologia já diagnosticada do paciente, podendo existir mais de uma para o
mesmo paciente. O tipo Tag relaciona fatores cotidianos que acontecem
com os pacientes, se ele bebeu ou dormiu mal na noite anterior, por exemplo.
Já o tipo HBI se refere ao Harvey Bradshaw Index, uma medida
padronizada para avalizar a gravidade da doença de Crohn,
especialmente.
|
|
trackable_name
|
Essa coluna descreve os tipos de
cada trackable_type, ou seja, o nome de cada Condition, Treatment,
Tag e de todos os outros elementos do tackable_type.
|
|
trackable_id
|
Demonstra um valor inteiro relativo
ao identificador único para cada “trackable_name”
|
|
trackable_value
|
Relata
um valor específico a cada trackable_name, mas de acordo com seu trackable_type.
Por exemplo, o grau de intensidade de determinado sintoma ou doença, assim
como a dose de um determinado tratamento.
|
Fonte: Próprio autor
Tendo
em vista essas descrições, nota-se que várias informações importantes para
futuras análises estão subdivididas em três principais colunas, a trackable_type,
trackable_name e trackable_value, onde dividem informações a
respeito de 7 categorias distintas, podendo cada uma ter uma coluna específica,
se dividindo apenas entre seus nomes e valores. Visto essa estrutura inicial, foi
necessário realizar a reestruturação da base através da criação de novas
colunas e agrupamento de usuários, descritos na subseção a seguir.
3.2
Pré-processamento
Tendo
em vista a má distribuição das informações observadas na análise descritiva, se
fez necessário modificar a estrutura original da base. No caso em questão, o
conteúdo da coluna trackable_type foi transformado em novas dimensões,
logo, foram criadas as colunas Condition, Symptom, Treatment, Weather, Tag,
Food e HBI. Seguindo a mesma lógica, também foram criadas colunas
referentes aos valores mapeados na coluna trackable_value, sendo elas: Condition_value,
Symptom_value, Treatment_value e Weather_value. As colunas Tag, Food e
HBI não possuem um valor específico associado aos seus registros.
Para
essa nova estrutura, além da criação das novas dimensões, foram feitas remoções
de registros com valores NaN, com idades superiores a 117 anos e menores que 0.
Também foram realizados agrupamentos utilizando as colunas user_id e checkin_date,
para pegar os registros de cada paciente a cada dia. Isso assegura, por
exemplo, que determinado sintoma foi registrado no mesmo dia de determinada
doença, ou que o mesmo sintoma esteja relacionado a algum fator cotidiano. A Figura 2 ilustra a transição da base
inicial para sua primeira modificação.
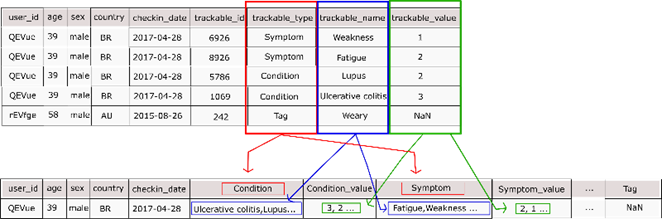
Figura 2: Reestruturação inicial da base Chronic
illness. Fonte: Próprio autor.
Após
esse procedimento, a base foi separada em 6 dataframes distintos, cada
uma possuindo o valor referente a um dos trackable_type da base
anterior, agrupando as informações do paciente (id, idade, sexo, data de
inserção e país) ao trackable_type com o valor “Condition”, que
agora será utilizado como coluna de target para futuro uso em
classificações supervisionadas.
Por
conseguinte, para a construção do modelo, se decidiu utilizar os dados
referentes aos sintomas dos pacientes, por estarem diretamente relacionados as
principais características descritivas das patologias relatadas.
Com o dataframe
apenas com as informações dos sintomas agrupadas, surgiu a necessidade de
separar as informações dos sintomas de cada paciente, pois agora existia uma
lista de sintomas em uma mesma coluna, podendo trazer complicações no momento
da inserção das informações em um futuro processo de classificação.
Por
esse motivo, foi criado uma nova coluna com o nome dos primeiros 250 sintomas
mais relatados entre os pacientes. Algumas variações dessa quantidade de
colunas foram realizadas, mas por limitações de hardware, 250 colunas foi o
maior valor suportada para o novo formato do dataframe.
Com as
novas colunas já criadas, foi adicionado o valor 1 quando o sintoma estava
relacionado ao paciente e o valor 0 quando esse valor não estava entre sua
lista de sintomas e, depois do agrupamento já realizado, foram eliminadas as
colunas user_id, checkin_date e country,
consideradas desnecessárias para o processo de predição. O resultado final da
base de sintomas é representado de forma resumida na Figura
3.
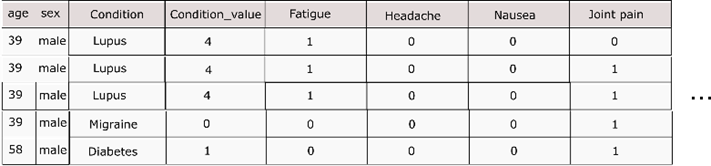
Figura 3: Resultado
do pré-processamento da base de dados, utilizando apenas os valores dos
sintomas. Fonte: Próprio autor.
Além
do exposto, houve também um processo de normalização e balanceamento das
informações através do recurso Categorical da biblioteca pandas
(utilizada para transformar o campo sexo em valores numéricos), factorize
(utilizado para fazer uma rotulação e categorização dos targets), StandardScaler
e o Normalizer do sklearn.preprocessing, para escalonar e
normalizar o restante dos dados [7]. Para balancear os dados foi utilizado um
processo denominado de Oversampling, deixando as amostras com
quantidades equivalentes, evitando uma futura classificação tendenciosa para as
classes com maior representatividade [8].
Por
fim, a base resultado de todo esse processo ficou com 254 colunas e um conjunto
de 300.884 instâncias relativas ao cruzamento dos sintomas com cada patologia.
Dessa forma, as informações se encontram melhor estruturadas para o uso em
futuras tentativas de classificação ou agrupamento de novas informações
3.3
Seleção do modelo
Para o
processo de seleção do modelo foram testados dois tipos diferentes de
abordagens. A primeira utilizando modelos de aprendizado de máquina [9] com o KNN e MLP e, a segunda utilizando
várias técnicas de ensemble a partir de um conjunto heterogêneo de
classificadores [10]. O melhor resultado é selecionado e
utilizado como modelo final. Nos tópicos seguintes, os modelos testados são detalhados.
3.3.1
Multilayer Perceptron (MLP)
A MLP [11] é um tipo de modelo que tem sido bastante
utilizado em problemas de auxílio ao diagnóstico de doenças, como relata Setsirichok et al. [12] e possui um bom comportamento em
problemas de múltiplas classes, segundo o mesmo autor.
Para o
problema em questão, a rede criada possui 4 vezes a dimensão de colunas da base
– 1 ((input_dim*4) -1) e 4 camadas escondidas com 128 neurônios cada
uma, utilizando a relu como função de ativação. Esses parâmetros foram
definidos através de um conjunto inicial de testes. Com uma estrutura de
camadas definida, foram testadas 4 combinações de épocas e batch size,
cada uma executada por 10 vezes (Tabela 1).
Tabela 1: Acurácia e taxa de perda de variadas
configurações de épocas e batch size para MLP com 10 classes
|
Número de Épocas
|
Batch Size
|
Taxa de perda
|
Acurácia
|
|
20.000
|
1.000.000
|
[0,09714625]
|
[0,71127867]
|
|
6.000
|
20.000
|
[0,07840306]
|
[0,72679924]
|
|
1.000
|
10.000
|
[0,10821427]
|
[0,69504005]
|
|
500
|
5.000
|
[0,11439618]
|
[0,73274235]
|
Fonte: Próprio autor
Analisando
os dois melhores resultados, através da visualização dos gráficos da Figura 4 percebe-se que o modelo do gráfico ‘A’
acaba entrando em overfitting[1] e se ajusta muito rápido ao
processo de aprendizado, sendo uma abordagem a ser descartada para a seleção do
modelo final. Por conseguinte, de todas as alternativas testadas a com 500
épocas e batch size de 5.000, obteve a melhor média da acurácia, com
aproximadamente 73,27 %.
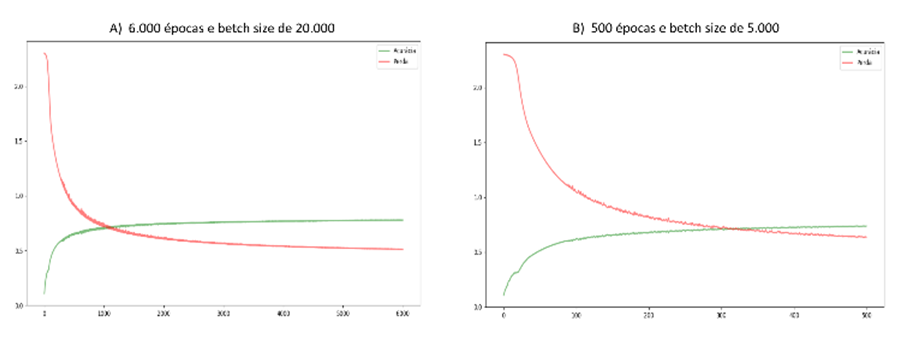
Figura 4: Gráfico
de convergência dos dois melhores resultados da MLP. Fonte: Próprio autor.
3.3.2
K-nearest neighbors (KNN)
classifier
O KNN
realiza seu processo de identificação de uma classe desconhecida a partir do
voto da maioria dos vizinhos mais próximos [13].
Nesse
projeto, todos os parâmetros desse modelo foram definidos por meio da execução
de um algoritmo genético (AG), onde o mesmo buscou encontrar a melhor
combinação dos parâmetros por meio de um processo baseado na escolha da melhor
solução [14] utilizando a acurácia do modelo como
função objetivo e variando os parâmetros relativos ao número de vizinhos (n_neighbors
ou K) e o tipo de algoritmo utilizado (algorithm). Como
resultado da seleção dos parâmetros, a melhor solução definiu o n_neighbors=2
(escolhendo de 1 a 10) e o algorithm = ‘kd_tree’ (escolhendo entre 'ball_tree','kd_tree','brute'
e 'auto').
Para a
evolução do modelo e divisão do conjunto de dados em treinamento e teste foram
utilizadas técnicas bastante comuns em aprendizado de máquina, como o TrainTestSplit
e a validação por meio de K-folds cross validation [15]. Seguindo o padrão de execução da MLP
(3.3.1), foram executadas 10 repetições com TrainTestSplit e 10
utilizando cross validation, variando o K-fold em 4, 7 e 10, como
é detalhado na Tabela 2.
Tabela 2: Resultados da média de 10 iterações do TrainTestSplit
e Cross Validation com variação de K-Fold de 4, 7 e 10 para o KNN
|
Divisão de base
|
Desvio Padrão
|
Acurácia
|
|
TrainTestSplit
|
[0,00310232]
|
[0,66935870]
|
|
Cross Validation: K-Fold=4
|
[0,00133886]
|
[0,67268614]
|
|
Cross Validation: K-Fold=7
|
[0,00104294]
|
[0,67855559]
|
|
Cross Validation: K-Fold=10
|
[0,00096968]
|
[0,68066290]
|
Fonte: Próprio autor.
3.3.3
Métodos Ensemble
Segundo
Chaudhary et al. [16] ensembles (veja mais sobre ensembles
em [10]) vêm sendo bastante recomendados para
problemas de classificação de várias classes. Tendo em vista isso, foram feitos
vários testes utilizando diferentes tipos de ensembles e diferentes quantidades
de classes para o problema em questão.
Inicialmente,
um conjunto de 10 classificadores foram executados separadamente na base de
dados utilizando seus parâmetros padrão. Dentre esses, os 4 que obtiveram a
melhor acurácia foram selecionados (RandomForestClassifier, KNeighborsClassifier,
ExtraTreesClassifier e DecisionTreeClassifier) para formar a base
dos classificadores do ensemble. O primeiro método executado foi o Max
Voting, que realiza uma votação entre as saídas de cada classificador
utilizado, tendo como saída a classe “mais votada” entre os classificadores
base [17].
Em uma
segunda abordagem de ensemble, foi utilizando o método Staking.
Ele utiliza o resultado da base de modelos do ensemble (camada 1)
como entrada para um modelo final (camada 2) [18]. Nesse tocante, surge as outras duas
técnicas de ensemble utilizadas, o Bagging e o Boosting
(para ler mais sobre, veja [18]).
Com base nessas
informações, nesse projeto tanto o Boosting como o Bagging foram
utilizados como modelos finais (camada 2) do Staking, com os mesmos classificadores
base do Max Voting (camada 1). Desse modo, dois foram os classificadores
utilizados para os testes com Boosting, o XGBoostClassifier e o CatBoostClassifier
[19]. Para os testes com Bagging foi
utilizado apenas o RandomForestClassifier. A
Tabela 3 demonstra
a média e desvio padrão da acurácia para 10 iterações utilizando 20, 10, 5, 3 e
2 classes para cada abordagem escolhida.
Tabela 3: Resultados de 10 iterações para os ensembles
dos 4 melhores classificadores utilizando diferentes números de classes
|
Método
|
Classificadores
|
Acurácia para 20 classes
Média
(Desvio Padrão)
|
Acurácia para 10 classes
Média
(Desvio Padrão)
|
Acurácia para 5 classes
Média
(Desvio Padrão)
|
Acurácia para 3 classes
Média
(Desvio Padrão)
|
Acurácia para 2 classes
Média
(Desvio Padrão)
|
|
Max Voting
|
Random Forest
KNeighborsClassifier
ExtraTreesClassifier
DecisionTreeClassifier
|
0,71455093
(0,00015715)
|
0,73728597
(0,00037731)
|
0,70891455
(0,00062014)
|
0,67871588
(0,00115697)
|
0,85081484
(0,00114085)
|
|
Staking
|
ExtraTreesClassifier
|
0,71540214
(0,00000000)
|
0,73717798
(0,00000000)
|
0,71037543
(0,00000000)
|
0,68302899
(0,00000000)
|
0,85156097
(0,00000000)
|
|
Staking /Bagging
|
Radom Forest
|
0,71514687
(0,00000000)
|
0,73778668
(0,00000000)
|
0,71127867 (0,00000000)
|
0,68237450 (0,00000000)
|
0,85156097
(0,00000000)
|
|
Staking /Boosting
|
XGBoostClassifier
|
0,71632501
(0,00000000)
|
0,73870955 (0,00000000)
|
0,71159284 (0,00000000)
|
0,68309444 (0,00000000)
|
0,85156097
(0,00000000)
|
|
CatBoostClassifier
|
0,71160265 (0,00000000)
|
0,73886664 (0,00000000)
|
0,71300660 (0,00000000)
|
0,68289810 (0,00000000)
|
0,85156097 (0,00000000)
|
Fonte: Próprio autor.
Dado a
variação dos valores médios das acurácias para o conjunto de testes com mais de
3 classes, notou-se que os classificadores poderiam estar se confundindo muito
em relação a algumas classes em específico, visto que ele sobe de ≈ 68% nos
testes com 3 classes para ≈ 85% nos testes com 2 classes, variando bastantes
nos testes anteriores. Desse modo, foi gerado uma matriz de confusão (Figura 5) dos
resultados obtidos com os esperados (targets) para melhor entender o
comportamento dos classificadores.
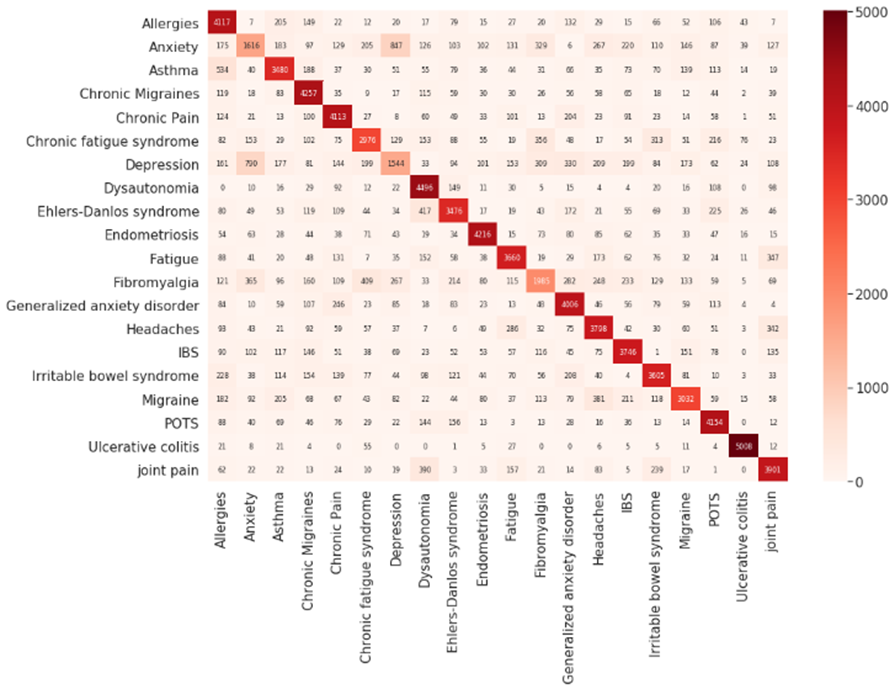
Figura 5: Matriz de confusão para o ensemble com
20 classes utilizando o XGBoostClassifier como classificador final do Staking.
Fonte: Próprio autor.
Analisando
a matriz de confusão, nota-se frequência mais alta de erros do classificador ao
tentar realizar a predição das classes “Depressão” e “Ansiedade”, podendo esse
ser o motivo da volatilidade dos resultados para as diferentes quantidades de
classes, visto que a escolha das classes é feita por sua maior
representatividade na base, onde “Depressão” e “Ansiedade” são a segunda e
terceira com maior número de amostras. A fim de evitar esse problema, foi
criado uma nova base, agora com a união dos dados referentes a ansiedade e
depressão, transformando-os em uma única classe. O resultado dessa nova
abordagem é demonstrado na Tabela 4.
De
acordo com os novos resultados, é perceptível uma normalidade maior em relação
à média da acurácia para seu conjunto de classes, tendo em vista que, quanto
maior o número de classes mais difícil será o processo de predição para o
classificador. Essa nova abordagem também melhorou bastante a média geral da
acurácia para cada conjunto de testes, crescendo em mais de 10% em alguns
casos.
3.4
Chronic Illness Diagnosis Helper (CIDH): definição da ferramenta
A
construção da prova de conceito foi inspirada em alguns dos aspectos do Design
Thinking, desenvolvido por Tim Brown [20]. O método proposto por Brown
Tabela 4: Resultados
de 10 iterações unindo as classes depressão e ansiedade para os ensembles dos 4
melhores classificadores utilizando diferentes números de classes.
|
Método
|
Classificadores
|
Acurácia para 20 classes
Média
(Desvio Padrão)
|
Acurácia para 10 classes
Média
(Desvio Padrão)
|
Acurácia para 5 classes
Média
(Desvio Padrão)
|
Acurácia para 3 classes
Média
(Desvio Padrão)
|
Acurácia para 2 classes
Média
(Desvio Padrão)
|
|
Max Voting
|
Random Forest
KNeighborsClassifier
ExtraTreesClassifier
DecisionTreeClassifier
|
0,76304991
(0,00009863)
|
0,80166618
(0,00018383)
|
0,83909919
(0,00060233)
|
0,83039136
(0,00048337)
|
0,86534925 (0,00063078)
|
|
Staking
|
ExtraTreesClassifier
|
0,76339764
(0,00000000)
|
0,80298158
(0,00000000)
|
0,84112833
(0,00000000)
|
0,83290029
(0,00000000)
|
0,86687778
(0,00000000)
|
|
Staking /Bagging
|
Radom Forest
|
0,76321495
(0,00000000)
|
0,80292069
(0,00000000)
|
0,84117704
(0,00000000)
|
0,83310328
(0,00000000)
|
0,86687778
(0,00000000)
|
|
Staking /Boosting
|
XGBoostClassifier
|
0,76379348
(0,00000000)
|
0,80310338
(0,00000000)
|
0,84151807
(0,00000000)
|
0,83298149
(0,00000000)
|
0,86687778
(0,00000000)
|
|
CatBoostClassifier
|
0,76579095
(0,00000000)
|
0,80362711
(0,00000000)
|
0,84190782
(0,00000000)
|
0,83294089
(0,00000000)
|
0,86626880
(0,00000000)
|
Fonte: Próprio autor.
aborda o design centrado
no ser humano, de tal forma que sua preocupação inicial deve ser explorar as necessidades
do usuário e, apenas depois criar o design do produto. Esse processo segue um
conjunto de três passos principais, a “inspiração” a “ideação” e por fim, a
“implementação” da ferramenta [21].
Para
esse projeto, a fase de “inspiração” é explorada na introdução desse documento,
através das justificativas para atacar o problema da dificuldade do diagnóstico
das DCNT e, a consequente necessidade da criação de uma ferramenta que pudesse
auxiliar na redução do tempo levado nesse processo. A etapa de ideação foi realizada
por meio da criação de um Mockup, validado por um conjunto de
profissionais de computação. A fase de “implementação” será abordada no tópico
a seguir.
3.5
Implementação da ferramenta
O desenvolvimento
da PoC final foi iniciado tendo como foco plataformas web, implementada com
base no framework Django e utilizando Bootstrap, JavaScript, HTML
e CSS como ferramentas para manipulação de suas propriedades gráficas. Todo layout
foi criado com a preocupação de manter um nível adequado de responsividade
sobre as telas.
Em
relação a base de dados, a aplicação contém as entidades usuarios, sintomas,
tem_paciente, tem_sintomas e pacientes. Dentre todas as
entidades, as que de fato estão sendo utilizadas no momento são sintomas e usuarios,
armazenando os 250 sintomas utilizados como colunas da base tratada e, os dados
dos médicos que utilizaram a aplicação, respectivamente.
Para
as telas da aplicação, a implementação foi feita com base no Mockup e
nas sugestões dos profissionais que participaram do processo de validação. A Figura 6 (A) demonstra como ficou a tela inicial.
Na Figura 6 (B) é demonstrada a tela de registro de
informações sobre o paciente.
Sugerido
no processo de validação, a tabela de fatores cotidianos foi substituída, dando
espaço a um campo interativo de pesquisa e uma listagem livre dos sintomas
selecionados. Ao fim da página existe também um botão para
dar início ao processo de predição do classificador. A tela de loading
utilizada durante a execução desse processo Figura 6(C) contém um gif com os ícones utilizados
na logo da ferramenta.
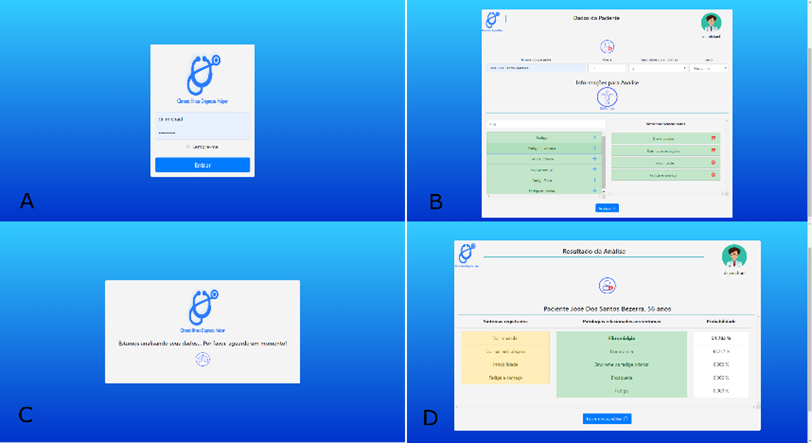
Por fim,
na Figura 6 (D) está a tela com os
resultados da análise feita pelo modelo, relatando além das doenças e da
probabilidade de cada uma, os sintomas utilizados como base para a predição. Ao
fim da página existe a opção de solicitar uma nova análise e, no canto superior
esquerdo das telas de resultado e registro de pacientes, há um ícone de um
médico, indicando a possibilidade de sair do sistema.
4
Discussões
Tendo
em vista as três principais fases do projeto, algumas considerações podem ser
feitas a respeito dos resultados obtidos por cada uma. Na etapa de manipulação
dos dados houve um grande esforço para que os dados pudessem ser organizados de
modo a proporcionar um conjunto de dados viável para uma futura classificação.
O fato das principais informações relativas as patologias dos pacientes estarem
todas em uma só coluna, dificultou bastante o processo de cruzamento das
informações.
Nesse
sentido, o ideal seria que cada amostra contivesse todas as informações
relativas a coluna trackable_type da tabela, ou seja, de uma única vez o
paciente inserisse informações sobre o tipo de sua doença, quais sintomas
estariam relacionados àquela doença, quais fatores (tags) interferiram
na intensidade dos sintomas, alimentação e clima, por exemplo, todos em uma
única amostra.
No
caso de tentar realizar essa união para a base em questão, haveria uma grande
perda de informações. Grande parte das amostras seriam eliminadas, podendo
trazer prejuízos para o processo de classificação, tendo em vista também o
crescimento das dimensões do problema.
Na
seleção dos modelos, cada abordagem foi testada com 10 iterações, salvando ao
fim a média e desvio padrão da acurácia de cada uma. A Figura 7 demostra os resultados referentes a
cada modelo, todos relativos a base com 10 classes
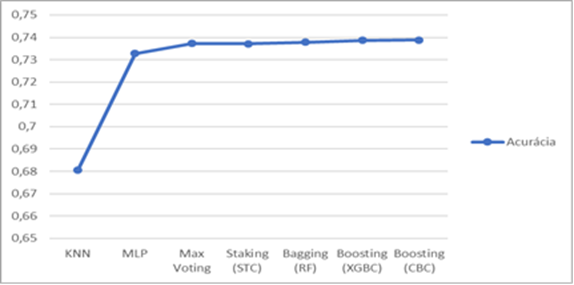
Figura 7: Média da acurácia de todos os modelos
testados para 10 classes. Fonte: Próprio autor.
Fica
perceptível uma certa similaridade entre as médias finais de cada modelo,
ficando apenas o KNN um pouco abaixo dos demais. Visto que os melhores
resultados foram os ensembles, mais especificamente o Boosting utilizando
CatBoostClassifier com ≈ 78,88 %, uma nova abordagem foi testada
para identificar possíveis melhorias para o processo de classificação.
Na
primeira abordagem, foi identificado através da análise da matriz de confusão,
uma tendência no processo de classificação. Haviam muitos erros dos modelos ao
tentar diferenciar as classes “Depressão” e “Ansiedade” e, por esse motivo,
elas foram unidas e os modelos foram novamente treinados com essa nova
configuração. De acordo com as duas abordagens, com e sem a união das classes, percebe-se
um crescimento de mais de 10% em alguns casos, sendo o melhor resultado para
duas classes, obtidos pelo ExtraTreesClassifier, RandomForest e XGBoostClassifier
com ≈ 86,68% de precisão.
Todavia,
devido ao ensemble utilizar vários classificadores para obter sua
predição, o tamanho do modelo final fica muito grande, acima de 2Gb (no caso de
10 classes) e acima de 4Gb (no caso de 20 classes), aumentando
significativamente o tamanho do projeto. Em contrapartida, o MLP com maior
acurácia (≈ 73,27%) não chega a 5Mb, podendo ser uma melhor alternativa
dependendo da infraestrutura existente para implantação da ferramenta. O uso ou
não dos modelos com as classes unidas traria maior precisão para as predições,
entretanto, esse tipo de informação deve ser analisado por um especialista de
saúde.
Por
fim, a PoC foi desenvolvida, sendo ajustada conforme algumas sugestões dos
avaliadores e com a viabilidade de implementação de algumas funcionalidades.
5
Conclusões
Nesse
projeto foi proposto uma nova abordagem, com base na criação de uma ferramenta,
para dar suporte a profissionais de saúde no auxílio ao diagnóstico de doenças
crônicas por meio de um modelo de aprendizado de máquina aplicado a dados
sintomáticos de pacientes.
Durante
todo o processo de desenvolvimento foram encontradas algumas dificuldades, em
relação a tempo, hardware e a desestruturação da base para com os objetivos
principais do trabalho. Entende-se que, com a estrutura necessária e com tempo
abio, técnicas como grid search ou algoritmos genéticos poderiam ser
utilizados para encontrar uma melhor combinação de parâmetros para os modelos
desenvolvidos.
Outra
possibilidade para a base seria o uso de algoritmos não supervisionados, na
tentativa de encontrar os melhores agrupamentos para as patologias. Os mesmos
também poderiam ser utilizados para separar essas patologias em subgrupos
(doenças crônicas cardíacas, reumatológicas, etc.) e, a partir deles, uma
classificação mais específica poderia ser realizada, tentando predizer um grupo
menor de doenças em áreas mais especializadas.
Para
trabalhos futuros, pretende-se desenvolver um novo módulo para a aplicação, que
terá como objetivo fazer o registro de todas as informações relatadas pela base
utilizada, porém, de maneira mais estruturada e separando as patologias por
área de especialização, já pensando na construção de um novo modelo de
classificação que possa ser ainda mais eficiente que o desenvolvido neste
projeto, tudo isso sendo adaptado também para dispositivos móveis.
Visto
o apresentado, fica evidente que um vasto esforço foi realizado para a obtenção
de um conjunto positivo de resultados, tanto no desenvolvimento do modelo de
classificação quanto na implementação da aplicação final, atingindo os
objetivos iniciais do projeto. Todavia, entende-se que ainda existe um grande
leque de oportunidades em aberto, possibilitando novas pesquisas e o
desenvolvimento de soluções cada vez mais eficientes.
6 Agradecimentos
Queria
agradecer a minha família, amigos e minha namorada Andressa, por me darem os
motivos que preciso para alcançar todos os objetivos da minha vida. Aos meus
colegas de residência que deram imensas contribuições para que esse trabalho
pudesse ser desenvolvido. Ao meu orientador Anthony Lins e ao professor Carmelo
Bastos pelas grandes colaborações e dicas e, as instituições FITec/SECTI/CMA-Parqtel/UPE/FACEPE
por incentivar o desenvolvimento profissional e tecnológico no estado por meio
de projetos como esse.
7
Referências
[1] World Health Organization., Saving lives, spending less
A strategic response to noncommunicable diseases, p. 20, 2018.
[2] Ministério
da Saúde, Vigilância de Doenças Crônicas Não Transmissíveis (DCNT),
2018. [Online]. Available:
http://www.saude.gov.br/vigilancia-em-saude/vigilancia-de-doencas-cronicas-nao-transmissiveis-dcnt.
[Accessed: 22-Aug-2019].
[3] Huang, M. J. ;Chen, M. Y.; Lee, S. C. Integrating
data mining with case-based reasoning for chronic diseases prognosis and
diagnosis, Expert Syst. Appl., vol. 32, no. 3, pp. 856–867, 2007.
[4] Mezzatesta, S. et al. A machine
learning-based approach for predicting the outbreak of cardiovascular diseases
in patients on dialysis, Comput. Methods Programs Biomed., vol. 177,
pp. 9–15, 2019.
[5] ABDAR,
M. et al. A new machine learning technique for an accurate diagnosis of
coronary artery disease, Comput. Methods Programs Biomed., vol. 179,
p. 104992, 2019.
[6] Almansour N. A. et al., Neural
network and support vector machine for the prediction of chronic kidney
disease: A comparative study, Comput. Biol. Med., vol. 109, no.
April, pp. 101–111, 2019.
[7] Singh,
D.; Singh, B. Investigating the impact of data normalization on
classification performance, Appl. Soft Comput. J., no. xxxx, p.
105524, 2019.
[8] Kim, Y. g; Kwon, Y.; Paik, M. C. Valid
oversampling schemes to handle imbalance, Pattern Recognit. Lett.,
vol. 125, pp. 661–667, 2019.
[9] Bates, A.; Saldias, B. A comparison
of machine learning and logistic regression in modelling the association of
body condition score and submission rate, Prev. Vet. Med., vol. 171,
no. November 2018, p. 104765, 2019.
[10] Herbrich, R.; Graepel, T. Ensemble
Methods, Foundations and Algorithms. Taylor & Francis Group, LLC, Cambridge,
UK, p. 232, 2012.
[11] Gardner, M. W.; Dorling, S. R. Artificial
neural networks (the multilayer perceptron) - a review of applications in the
atmospheric sciences, Atmos. Environ., vol. 32, no. 14–15, pp.
2627–2636, 1998.
[12] Setsirichok, D. et al., Classification
of complete blood count and haemoglobin typing data by a C4.5 decision tree, a
naïve Bayes classifier and a multilayer perceptron for thalassaemia screening,
Biomed. Signal Process. Control, vol. 7, no. 2, pp. 202–212, 2012.
[13] Poddar, M. G. et al. Automated
Classification of Hypertension and Coronary Artery Disease Patients by PNN,
KNN, and SVM Classifiers Using HRV Analysis. Elsevier Inc., 2019.
[14] Parinam, S. et al. An improved
optical parameter optimisation approach using Taguchi and genetic algorithm for
high transmission optical filter design, Optik (Stuttg)., vol. 182,
pp. 382–392, 2019.
[15] Kajó, M.; Nováczki, S. A Genetic
Feature Selection Algorithm for Anomaly Classification in Mobile Networks, Proc.
19th Int. ICIN Conf.-Innov. Clouds, Int. Netw., pp. 204–211, 2016.
[16] Chaudhary, A.; Kolhe, S.; Kamal, R. A
hybrid ensemble for classification in multiclass datasets: An application to
oilseed disease dataset, Comput. Electron. Agric., vol. 124, pp.
65–72, 2016.
[17] Hosni, M. Reviewing ensemble
classification methods in breast cancer, Comput. Methods Programs
Biomed., vol. 177, pp. 89–112, 2019.
[18] Ribeiro, M. H. D. M.; Coelho, L. S. Ensemble
approach based on bagging, boosting and stacking for short-term prediction in agribusiness
time series, Appl. Soft Comput. J., p. 105837, 2019.
[19] Huang G. et al., Evaluation
of CatBoost method for prediction of reference evapotranspiration in humid
regions, J. Hydrol., vol. 574, no. December 2018, pp. 1029–1041,
2019.
[20] BROWN,
T. Change by design:How Design Thinking Transforms Organizations and
Inspires Innovation. HarperBusiness, 2009.
[21] Bastos, M.
L. CatalogToMakers :
uma plataforma de catalogação colaborativa de componentes eletrônicos e
projetos de computação física, Universidade Federal de Pernambuco, 2019.
[22]
Hawkins, D. M. The Problem of
Overfitting, J. Chem. Inf. Comput. Sci., vol. 44, no.
1, pp. 1–12, 2004.